‘네트워크 임베딩 기술’ 적용…정확도 93%
한양대학교 컴퓨터소프트웨어학부 서지원 교수팀이 울산과학기술원(UNIST) 이세민 교수팀, 고려대 정원기 교수팀과 함께 '다중 오믹스(Multi-Omics) 데이터 기반의 환자 맞춤형 항암제 반응성 예측을 위한 기계학습 모델'을 개발했다. 환자별로 항암제의 효능을 미리 알 수 있는 인공지능(AI) 기술이라는 점에서 의미가 크다. 똑같은 암을 앓는 환자라도 항암제의 효능은 달라질 수 있기 때문이다.
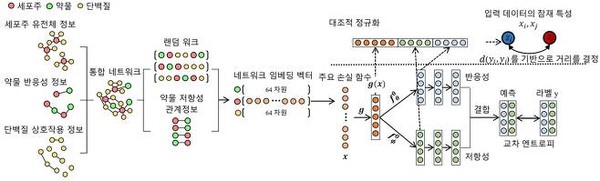
공동 연구진은 ‘네트워크 임베딩 기술’을 적용해 다차원 데이터 간 상관관계를 효과적으로 반영하여 기존 기계학습 모델보다 정확도를 높였다. 암세포에서 파생된 세포주와 항암제, 유전자를 ‘노드’(연결점)로 삼고, 각 노드를 연결해 ‘엣지’(연결선)를 만들었다. 특히 노드와 엣지로 형성된 네트워크 세트의 상관관계를 반영한 '임베딩 벡터'(embedding vector)의 추출은 이번 연구의 핵심이다. 임베딩 벡터를 이용하면 각 노드의 대푯값을 알 수 있어 고차원적인 데이터도 효과적으로 다룰 수 있다. 또 연구진은 임베딩 벡터를 인공지능 기법인 심층신경망으로 학습시켜 환자 맞춤형으로 항암제 효능을 도출했다. 연구진 설명에 따르면 개발된 모델의 항암제 반응성 예측 성능은 기존 모델보다 크게 향상된 93% 정도의 정확도를 기록했다.
한편 연구 결과는 생명정보학 분야 학술지인 '브리핑스 인 바이오인포메틱스'(Briefings of Bioinformatics)에 공개됐다.
관련기사
키워드
정연 커뮤니케이터
cky6279@hanyang.ac.kr


 '한양위키' 키워드 보기
'한양위키' 키워드 보기